使用 kallisto 可以对原始 RNA-seq 数据的 Reads 结果进行分析,统计每一基因的 counts 估计值、 TPM 值等对寻找差异表达基因有用的值, kallisto 是基于 pseudoalignment 的算法,能够快速分析二代测序结果,而其结果的分析与解读工具又可以使用相应的 R 包来完成,就是 Sleuth,不过这个包还再开发完善中,并未发表相关的文章,鉴于是针对 Kallisto 的下游分析工具,可以先来尝试一下
Sleuth 包的安装
- Sleuth 包还没有发布,所以只能通过 github 来安装,先安装
rhdf5再安装devtools最后通过 github 安装sleuth - 安装:
>source("http://bioconductor.org/biocLite.R")
>biocLite("rhdf5")
>install.packages("devtools")
>devtools::install_github("pachterlab/sleuth")
数据导入
- sleuth 是基于 kallisto 结果的后续分析,所以可以直接读取 kallisto 输出的结果
abundance.h5文件或者abundance.tsv文件 - abundance.tsv 结果如下:
target_id length eff_length est_counts tpm
1 ENST00000415118 8 5.0000 0 0
2 ENST00000448914 13 10.0000 0 0
3 ENST00000434970 9 6.0000 0 0
4 ENST00000390577 37 10.0625 0 0
5 ENST00000437320 19 16.0000 0 0
6 ENST00000450276 17 14.0000 0 0
- 下载Sleuth测试数据进行测试,放在
~/sample/kallisto/test/下
>library("sleuth")
>base_dir <- "~/sample/kallisto/test/"
>sample_id <- dir(file.path(base_dir,"results"))
>kal_dirs <- sapply(sample_id, function(id) file.path(base_dir, "results", id, "kallisto"))
>s2c <- read.table(file.path(base_dir, "hiseq_info.txt"), header = TRUE, stringsAsFactors=FALSE)
>s2c <- dplyr::select(s2c, sample = run_accession, condition)
>s2c <- dplyr::mutate(s2c, path = kal_dirs)
>so <- sleuth_prep(s2c, ~ condition)
reading in kallisto results
......
normalizing est_counts
50844 targets passed the filter
normalizing tpm
merging in metadata
normalizing bootstrap samples
summarizing bootstraps
分析及结果输出
>so <- sleuth_fit(so)
>so <- sleuth_wt(so, which_beta = 'conditionscramble')
>results_table <- sleuth_results(so, 'conditionscramble')
>write.csv(results_table,"results_test.csv")
> head(results_table)
target_id pval qval b se_b mean_obs var_obs
1 ENST00000075120 0 0 -1.4661181 0.03535490 7.910524 0.6463507
2 ENST00000244745 0 0 -1.6483482 0.04193705 7.568028 0.8172260
3 ENST00000249749 0 0 -2.6770431 0.06291763 6.452782 2.1533039
4 ENST00000256646 0 0 0.9454387 0.02496143 8.218254 0.2688586
5 ENST00000257497 0 0 1.0245592 0.02474028 8.313881 0.3156510
6 ENST00000263734 0 0 1.3233153 0.02526430 8.213073 0.5255182
tech_var sigma_sq smooth_sigma_sq final_sigma_sq
1 0.0006033334 1.271621e-03 0.0006840236 0.0012716205
2 0.0007067384 1.931335e-03 0.0007652357 0.0019313352
3 0.0042122927 -4.232682e-05 0.0017256493 0.0017256493
4 0.0002716565 6.062440e-04 0.0006629529 0.0006629529
5 0.0002418629 6.762591e-04 0.0006657409 0.0006762591
6 0.0002944993 -8.305197e-05 0.0006629280 0.0006629280
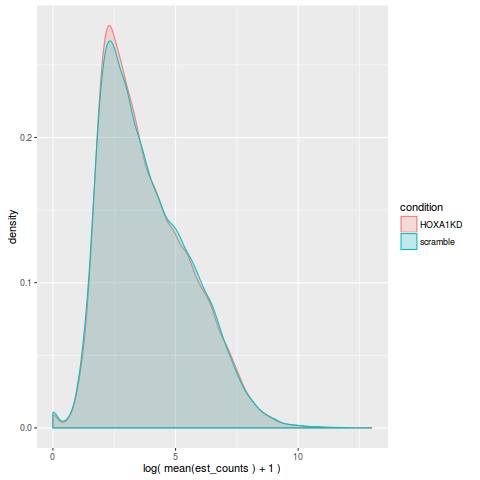
组间 counts 密度图
>plot_group_density(so)
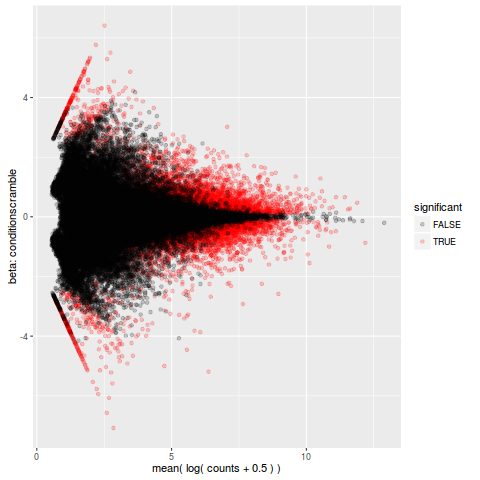
MA 图
>plot_ma(so,test='conditionscramble')
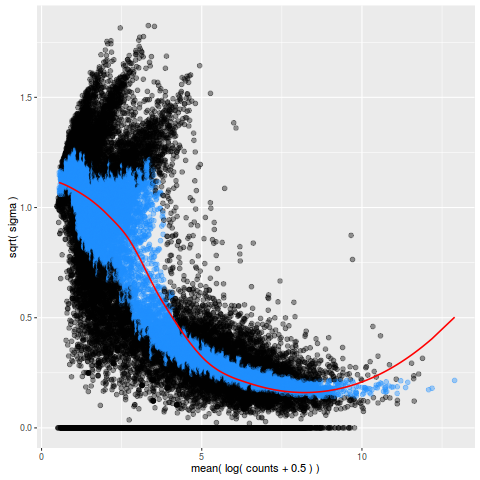
均值-方差分析
>plot_mean_var(so)
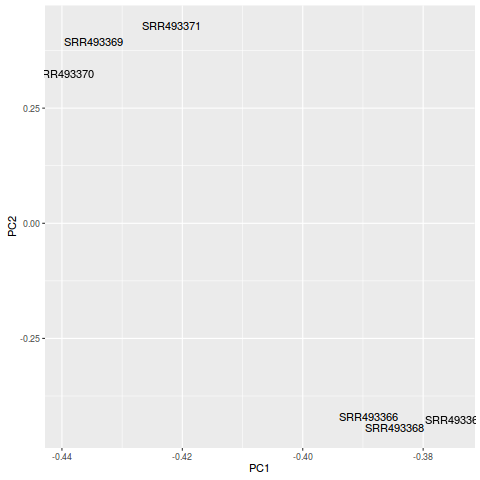
PCA分析
>plot_pca(so,text_labels=TRUE)
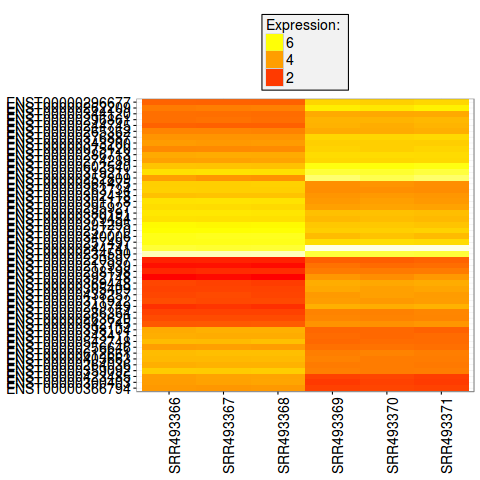
heatmap(top50) 图
>library(pheatmap)
>plot_transcript_heatmap(so,transcripts = results_table$target_id[1:50])
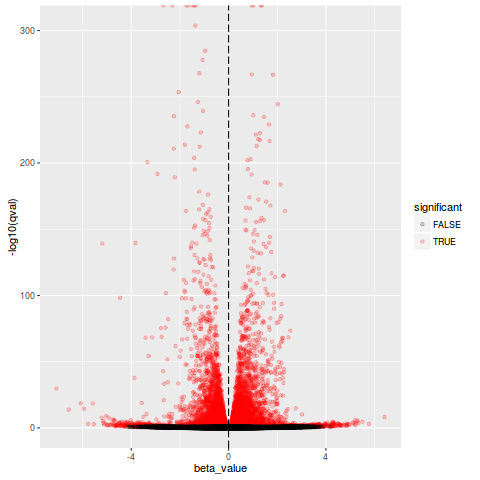
火山图
>plot_volcano(so,test="conditionscramble")